





|
遺伝情報の流れ(セントラルドグマ)によれば、DNAからRNAを経てタンパク質がつくられる。
転写 翻訳 DNA RNA タンパク質 RNA
polymeraseリボソーム
tRNA[DNAからタンパク質への流れ]
DNAからRNAを合成する段階を転写(transcription)という。RNAはDNAの−鎖(アンチセンス鎖)を鋳型として作られる。このとき,鋳型鎖のGはC,CはG,TはA,AはU(RNAではTの代りにU)と読み取られる。
5' -----GCA-AAT-TCC-GGT----- 3'
3' -----CGT-TTA-AGG-CCA----- 5'非鋳型鎖,情報(コード)鎖,センス鎖,+鎖
鋳型鎖,非コード鎖,アンチセンス鎖,−鎖転写 RNAポリメラーゼ
5'-----GCA-AAU-UCC-GGU----- 3' RNA [+鎖] DNAの鋳型鎖(−鎖,アンチセンス鎖)がRNA合成の鋳型となる
原核生物では転写と翻訳(タンパク質合成)は同時に進行する。解糖系の酵素群のように,生物の生存に必要な最小限のタンパク質の遺伝子(ハウスキーピング遺伝子)は常にスイッチONの状態にあるが,タンパク質合成は多くのエネルギーを消費するので,生物にとって無駄な遺伝子の転写や翻訳は避けなければならない。従って,多くの遺伝子のうち、どれを、いつ、どこで、どの程度発現させるかは、生物にとって最も重要な事のひとつである。外界からのさまざまな刺激や真核生物生物が元来もっている発生、分化、増殖、加齢などのプログラムによって、個々の遺伝子の転写は精妙に調節される。
転写を触媒する酵素はRNAポリメラーゼで、これには多くのタンパク質が関与する。原核生物の場合,プロモーターと呼ばれる特有のDNAの塩基配列がs因子で認識されて転写が開始される。真核細胞では転写制御因子が遺伝子の上流のエンハンサー配列に結合し,同時に,プロモーターを読み取ることによって転写が開始される。
  
|
DNAの鋳型鎖[−鎖]の塩基配列を読み取って相補的なRNAを合成する反応(転写)を触媒する中心となる酵素をDNA依存性RNAポリメラーゼ(以下,単にRNAポリメラーゼと呼ぶ)という。ヌクレオチド鎖の合成方法はDNAポリメラーゼの場合と似ているが,RNAポリメラーゼがプライマーを必要としないことは際立った違いである。なお,RNAポリメラーゼには,ある種のウィルスに見られるRNA依存性RNAポリメラーゼや,鋳型を必要としない真核生物のポリ(A)ポリメラーゼなどもある。
 大腸菌のRNAポリメラーゼ
大腸菌のRNAポリメラーゼ
| ヌクレオチドや RNAと結合 (触媒中心) |
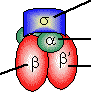 |
プロモーターを 読み取る。 bとb’を結合 DNAと結合 (Zn2+含有) |
 |
||
| [大腸菌のRNAポリメラーゼ (ホロ酵素)] 青と水色:a,青緑:b,赤:b'サブユニット |
||
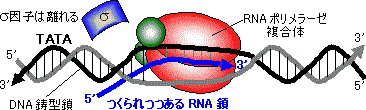 |
||
| [大腸菌RNAポリメラーゼによるRNA鎖の伸長] 合成速度は40ヌクレオチド/秒 |
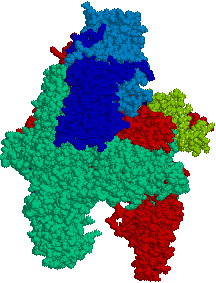
 真核生物のRNAポリメラーゼ
真核生物のRNAポリメラーゼ
 RNAポリメラーゼI: 核小体に存在。rRNA前駆体を合成。
RNAポリメラーゼI: 核小体に存在。rRNA前駆体を合成。 RNAポリメラーゼII: 核質に存在。hnRNA(mRNA前駆体)、snRNAを合成。
RNAポリメラーゼII: 核質に存在。hnRNA(mRNA前駆体)、snRNAを合成。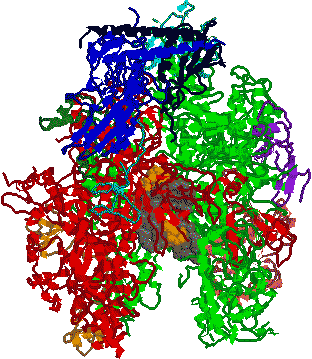
 RNAポリメラーゼIII: 核質に存在。tRNAと5S rRNAを合成。
RNAポリメラーゼIII: 核質に存在。tRNAと5S rRNAを合成。
  
|
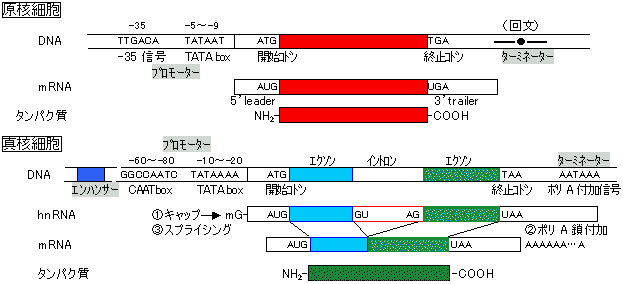
 原核生物(大腸菌)の場合
原核生物(大腸菌)の場合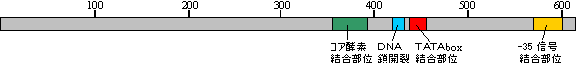 |
| s70因子の構造 |
また、転写を開始するためにはプロモーター配列を正しく認識することに加えて、転写開始点付近のDNA2本鎖を1本鎖にほどく必要があるが、σのDNA鎖開裂領域にある芳香族アミノ酸が、DNA塩基とのスタッキングによってDNAの1本鎖状態を安定化する。転写開始に関わる-10配列および-35配列をプロモーター(promoter)という。発現レベルの高い一部のプロモーターでは、-35配列よりさらに上流10〜30塩基の位置にATに富んだ配列が存在し、これによって転写活性が数十倍に増強されることが知られている。この第三の構成要素はUPエレメントと呼ばれる。UPエレメントはRNAポリメラーゼのαサブユニットのC末端ドメインによって認識される。
一方,転写の終了を調節する配列をターミネーター(terminater)という。これらの信号はシスに作用する(同じDNA鎖上で働く)のでシスエレメントという。
プロモーター
[大腸菌とバクテリオファージのs70プロモーターの例] 6種のs因子は別々のプロモーターを認識する。通常はs70が使われるが,条件によっては別のs因子も用いられる。例えば熱ショック時はs32。これにより,それぞれの環境において,異なるプロモーターをもつ遺伝子の発現を可能にしている。
s70 -10領域:TATAAT (Pribnow配列[TATA box])
-35領域:TTGACA (-35信号)
s32 -10領域:CCCCATNT (Nは任意)
-35領域:CCCTTGAA
s54 -12領域:TTGCA
-24領域:CTGGNA
s28 -10領域:GCCGATAA
-35領域:CTAAA- 転写は,(1) DNA鎖の巻き戻し,(2) プロモーターへのs因子/RNAポリメラーゼ複合体の結合,(3) s因子の遊離,(4) ポリメラーゼによる転写の開始と進行,(5) DNA鎖の巻き直し,の順に進む。
ターミネーター
1. 回文(パリンドローム)配列
G:Cに富む回文は強固なヘアピン構造をとり,この後ろにUが連続する。
このUと鋳型DNAのAは弱い水素結合で結びついているので,RNAは
鋳型から離れやすい。[大腸菌の回文型ターミネーター]
回文構造はループを形成する。2. 転写終結因子(r因子)の結合による転写終了もある。
r(ロー)因子はヘリカーゼ活性をもち,DNA-RNA間の水素結合を切る。オペレーター
ポリシストロン転写単位の遺伝子(オペロンという)の場合、転写はリプレッサータンパク質やオペレーター部位での制御を受ける。
大腸菌のラクトース(lac)オペロンの転写制御
プロモーターから構造遺伝子領域までがオペロンである。 グルコースが利用できる場合,lac オペロンのオペレーター部位にlac リプレッサータンパク質が結合し転写を抑制している。
もし,グルコースが枯渇し,代わりに乳糖が利用できる場合は,乳糖がリプレッサーに結合して不活性型に変換するため,リプレッサーはオペレーター部位を離れ,プロモーター部位が認識できるようになるため,転写が開始される。
[lac リプレッサータンパク質の構造] [lac リプレッサーとオペレーターの結合] 4量体タンパク質で,4つのサブユニットは図の上方のへリックスで
結びついている。DNAのオペレーター部位と結合するのはサブユニット
の端である。リプレッサーが結合すると,DNAは湾曲
し,TATA boxが読み取れなくなる。
大腸菌のトリプトファン(trp)オペロンの転写制御
細胞内のトリプトファン濃度が高い場合は,TrpはTrp リプレッサーに結合して活性型に
しているため,リプレッサーが転写を抑制している。このような作用をもつ分子をコリプレッ
サーという。[Trpと結合したTrp リプレッサー] もし,トリプトファン濃度が低くなるとTrpがリプレッサーから遊離するため,リプレッサーは
本来の不活性型に戻る(lac リプレッサータンパク質と逆である)。その結果,リプレッサーは
オペレーター部位を外れ,トリプトファン合成酵素遺伝子群の転写が開始され,必要な量の
トリプトファンが合成できるようになる。
 真核生物の場合
真核生物の場合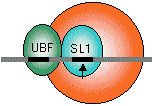 |
 |
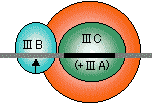 |
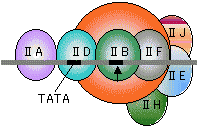
| RNAポリメラーゼI | RNAポリメラーゼII | RNAポリメラーゼIII |
| [真核細胞のRNAポリメラーゼ(基本転写因子)] | ||
| 灰色横線はDNAを、黒い部分はシスエレメントを表す。矢印は転写開始位置(+1残基)。 SL1, II D, III BはいずれもTBP (TATA Binding Protein)をサブユニットにもつ複合体である。 |
||
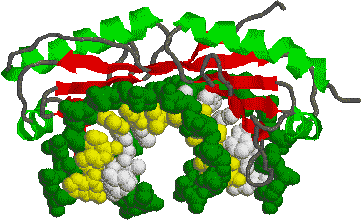 [(TATAボックス結合タンパク質] |
| TATA配列をもつDNA分子(主鎖を濃緑で,側鎖を白と黄色で示す)に上から結合したTATAボックス結合タンパク質。 |
 RNAポリメラーゼIの場合
RNAポリメラーゼIの場合 RNAポリメラーゼIIの場合
RNAポリメラーゼIIの場合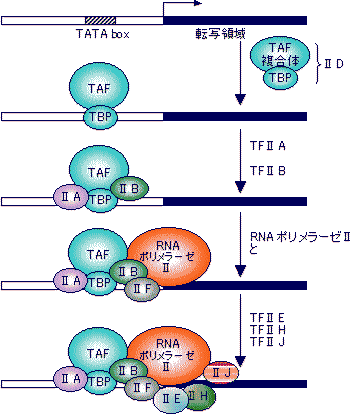
 RNAポリメラーゼIIIの場合
RNAポリメラーゼIIIの場合
  
|
 エンハンサー配列やサイレンサー配列
エンハンサー配列やサイレンサー配列| 応答配列(略語) | コンセンサス配列 | 結合因子 |
| グルココルチコイドRE(GRE) エストロゲン応答配列(ERE) 血清応答配列(SRE) 熱ショックエレメント(HSE) cAMP応答配列(CRE) TPA応答配列(TRE) p53応答配列 E2F応答配列 赤血球GATA応答配列 |
AGAACANNNTGTTCT AGGTCANNNTGACCT CCATATTAGG CNNGAANNTCCNNG TGACGTCA TGACTCA PuPuPuC(A/TNA/T)GPyPyPy TTTCGCGC GATA |
グルココルチコイド受容体(下図) エストロゲン受容体 Serum Response Factor Heat Shock Factor CREB(ATF) AP-1(Jun/Fos) p53(癌抑制遺伝子産物) E2F GATA-I |
 転写因子(transcription factor)
転写因子(transcription factor)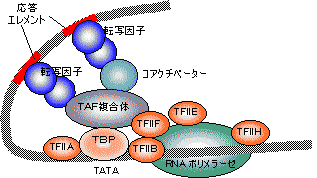 |
| [転写因子の作用様式] |
 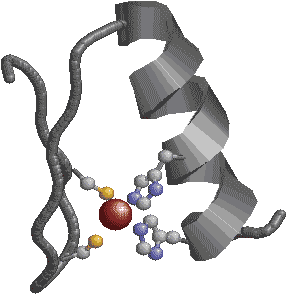 |
|
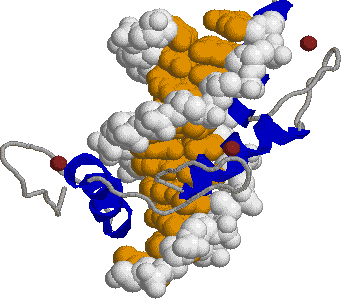
| [ジンク(Zn)フィンガー構造] | |
| (左)転写因子ZIF268。中央に黄色と白で示したDNA鎖の主溝にはまり込むようにして,転写因子の3つのジンクフィンガーが結合している。茶色の球はZnイオンである。 (右)His2-Cys2型Znフィンガー。2つのS原子と2つのN原子がZnイオンに正四面体状に配位している。Cys4型Znフィンガーもある。 |
 |
 |
|
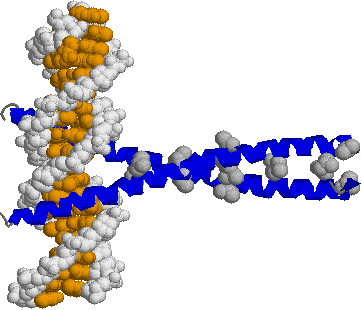
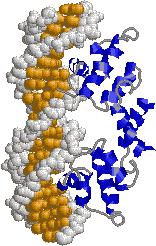
| [ロイシンジッパー構造] | [helix-turn-helix (HTH)構造] | |
| 巨大なへリックスがDNAを挟むようにして,主溝に結合する。ロイシン残基を赤で示す。 | HTH構造のそれぞれ1つのへリックスがDNAの主溝に結合する。 | |
 DNAのメチル化
DNAのメチル化
  
|
 イントロン (intron)
イントロン (intron) エクソンとイントロンの例
エクソンとイントロンの例| エクソン | イント ロン |
エクソン | イントロン | エクソン | ||
| 非転写領域 | 非転写領域 | |||||
| [ヒトb-グロビン遺伝子(genome DNA)の構成] | ||||||
| 転写領域は3つのエクソン(赤)と2つのイントロンで構成される。 | ||||||
 |
| [ヒトb-グロビン遺伝子の全ヌクレオチド配列] |
| 3つのエクソン部分を赤で、イントロン部分を黄色で示す。イントロンはGTで始まり、AGで終わる。非転写領域には,CAATボックスやTATAボックスがある。ポリA付加信号が転写領域の終わりの部分に見られる。蛋白質のアミノ酸配列に対応する核酸配列部分をオープンリーディングフレーム(open reading frame, ORF)という。図では,開始コドンから終止コドンの間がORFである。 |

 プロセシング(processing)
プロセシング(processing) キャップ構造の付加(キャッピング反応)
キャップ構造の付加(キャッピング反応)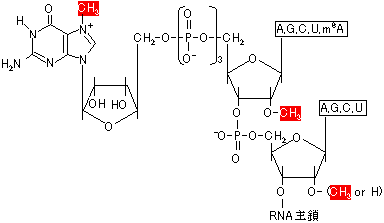 |
| [mRNAのキャップ構造] m7GpppがRNAの3'末端に結合。RNAの最初の2残基はメチル化。 |
 3'末端のポリ(A)鎖
3'末端のポリ(A)鎖 スプライシング
スプライシング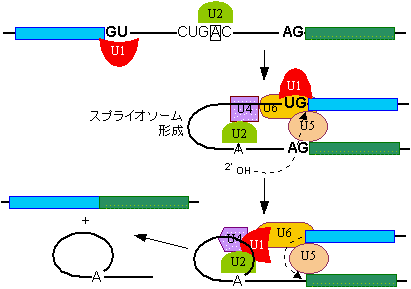 |
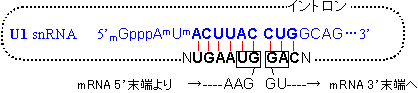 |
| [snRNAsによるRNAのスプライシング機構] |
| 《hnRNA以外のRNAのスプライシング》 tRNAやrRNAの場合は、GU-AG則は当てはまらず、スプライシングの仕方は上とは異なる。 |
 選択的スプライシング(alternative splicing)
選択的スプライシング(alternative splicing)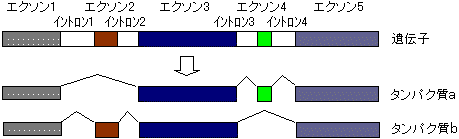
  
|
 ヘテロ核RNA(HnRNA、真核生物のみ)
ヘテロ核RNA(HnRNA、真核生物のみ)
 伝令RNA(mRNA)
伝令RNA(mRNA)
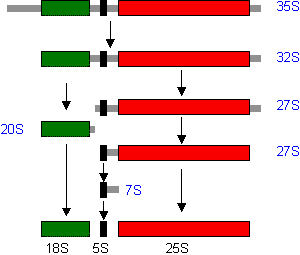
 リボソームRNA(rRNA)
リボソームRNA(rRNA)
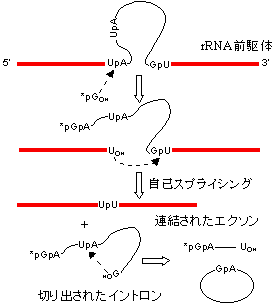
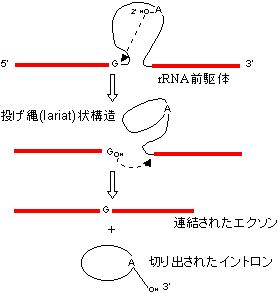
[(A)グループI型イントロンの除去(テトラヒメナrRNAの自己スプライシング)]
グアノシンとMg2+(or Mn2+)が必須。(B)グループII型イントロンの除去。ポリアミンを補助因子として要求。
 転移RNA(tRNA)
転移RNA(tRNA)
  |
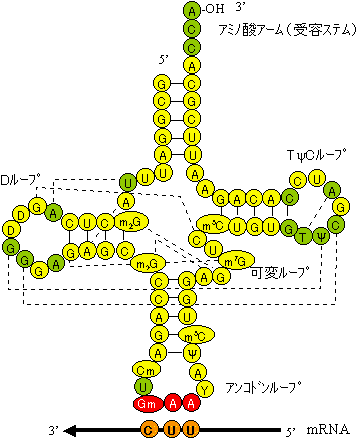 |
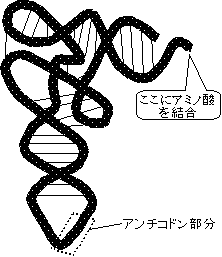
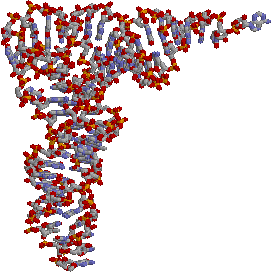
| [酵母フェニルアラニンtRNA(tRNAPhe)の1次および2次構造(右)と3次構造(左)] | |
| クローバー葉形に並べた塩基配列。緑色の丸は全てのtRNAで保存されている残基。破線は立体相互作用塩基対。m2G, D, Cm, Gm, Y, y, m5G, m7G, m5Cは特殊塩基。 | |
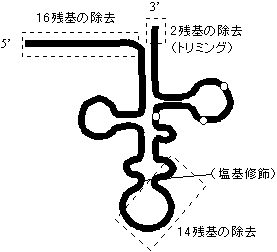 |
プロセシング |
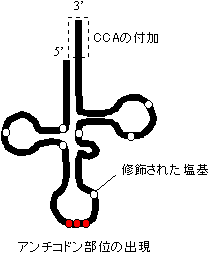 |
| [酵母Tyr-tRNAのプロセシング] | ||
 ミトコンドリアRNA(真核生物)
ミトコンドリアRNA(真核生物)
 snRNA(small nuclear RNA、真核生物)
snRNA(small nuclear RNA、真核生物)
 トランスファーメッセンジャーRNA(tmRNA、原核生物)
トランスファーメッセンジャーRNA(tmRNA、原核生物)
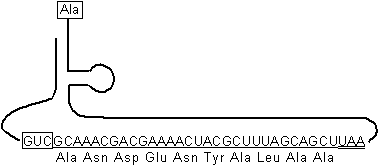
[大腸菌ssrA RNA]
枠内に対応するmRNAが無くてもAlaを付ける。下線部はタグ配列。二重下線は終止コドン。
 その他のRNA: 低分子RNA(scRNA、snoRNA)など。
その他のRNA: 低分子RNA(scRNA、snoRNA)など。
| 《自己切断を起こすRNA(ribozyme)》 1) ウイロイド(viroid) 殻をもたない裸の一本鎖、環状、低分子RNA。ジャガイモやせいも病など植物の病原体。 2) ウィルソイド(virusoid) 一本鎖、環状RNA。直線状RNAウィルスと一緒に粒子中に存在。単独では感染性なし。 これらはともにローリングサークル型で複製される。また、自己触媒的にプロセシングを起こす。構造上、次の2つがある。 1. ハンマーヘッド型リボザイム 2. ヘアピン型リボザイム 3) その他 T4ファージRNA前駆体、d型肝炎ウィルスRNA、アカパンカビのミトコンドリアのプラスミド転写物なども自己切断や自己連結を起こす。 |
 (エディティング) =>セントラルドグマへの挑戦 (エディティング) =>セントラルドグマへの挑戦
|
  |
タンパク質の1次構造は全てDNAが決定すると信じられてきた。つまり、DNAの情報を写しとり、不要な部分を切りすて、忠実にタンパク質の情報に置き換えるのがRNAの役割である。しかしながら、この遺伝情報の流れ(セントラルドグマ)に挑戦するかのような現象が、トリパノソーマ(原鞭毛虫類、睡眠病の原虫)のキネトプラスチドDNAの転写過程に見出された。そこではウリジンの挿入や欠失が見出され、つくられるタンパク質の1次構造が変えられていた。
トリパノソーマ・キネトプラスチド(ミトコンドリアに相当)のシトクロムcオキシダーゼ遺伝子(CO
III)mRNA(731塩基)において、145ヶ所で計407個のウリジンが挿入され、9ヶ所で計19個のウリジンが欠失していた。このようにmRNAの塩基配列を変えることにより、翻訳されるタンパク質に変化をもたらす現象をRNAエディティング(RNA
editing)と呼ぶ。RNA編集はプロセシングの1種である。
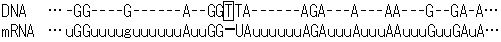
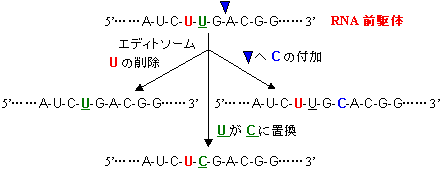 |
| [RNA編集のタイプ] |
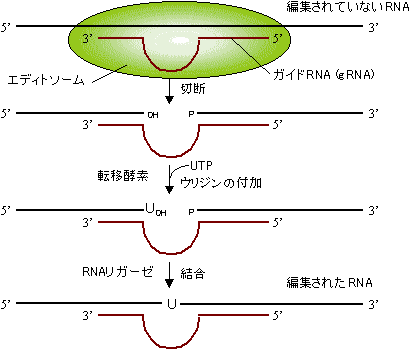 |
| [トリパノソーマmRNAの編集機構] |
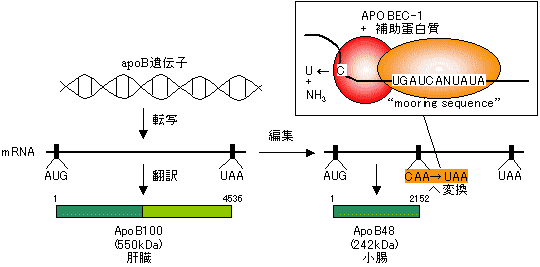 |
| [アポリポタンパク質mRNAの小腸における編集] |
 RNA編集の意義
RNA編集の意義
 「センスコドン<=>終止コドン」の変化で翻訳産物の大きさが変化
「センスコドン<=>終止コドン」の変化で翻訳産物の大きさが変化 塩基挿入によりフレームシフトが起き、翻訳産物の大きさや分子後半の配列が変化
塩基挿入によりフレームシフトが起き、翻訳産物の大きさや分子後半の配列が変化 特定のアミノ酸のみが置換され、翻訳産物の機能が変化
特定のアミノ酸のみが置換され、翻訳産物の機能が変化 塩基配列の変化により、RNAの安定性などが変化
塩基配列の変化により、RNAの安定性などが変化
| トリパノソーマでは、その生活環に合わせてミトコンドリアの機能が大きく変化する。発生の分化の段階によってmRNAの編集を行えば、構造的、機能的に異なる蛋白質をつくることが可能となる。 |
| 種類 | 標的RNA | 触媒 | 結果 |
| キャップ付加 ポリ(A)付加 スプライシング トリミング 塩基修飾 編集 |
HnRNA HnRNA HnRNA, pre-rRNA, pre-tRNA 全RNA pre-tRNA mRNA |
キャップ形成酵素 ポリ(A)ポリメラーゼ スプライセオソーム またはpre-RNA自身 ヌクレアーゼ 特定の酵素 エディトソーム |
5'にキャップが付加される 3'にポリ(A)鎖が付加される イントロンが除去される ヌクレオチド残基が3'末端から除去される 特定の塩基が特殊塩基に変わる ヌクレオチド残基が付加、削除、変更される |
|
 原核生物に作用するもの
原核生物に作用するもの| 阻害剤 | 作用 |
| アクチノマイシンD リファマイシンと リファンピシン ストレプトリジギン |
二本鎖DNAの隣り合うG:C塩基対間にはまり込む。 RNAポリメラーゼを阻害。 RNAポリメラーゼのbサブユニットに結合し、転写の開始を阻害する (RNA鎖の延長は阻害しない。) RNA鎖の延長を阻害する(転写の開始は阻害せず)。 |
 真核生物に作用するもの
真核生物に作用するもの| 阻害剤 | 作用 |
| アクチノマイシンD a-アマニチン |
二本鎖DNAの隣り合うG:C塩基対間にはまり込む。 RNAポリメラーゼを阻害。 RNAポリメラーゼIIを強く阻害。RNAポリメラーゼIは阻害せず。 RNAポリメラーゼIIIは高濃度で阻害。 |

|