








       
|
 (DNA polymerase) (DNA polymerase) |
 |
 |
[DNA�|�������[�[�̓����̂܂Ƃ�]
|
 �咰�ۂ̍y�f
�咰�ۂ̍y�f
| ���� | I | II | III |
| ���q����(kD) �T�u���j�b�g ���q��/�זE �������x(NTP/��) �\����`�q ��������: 5'��3' ��������: 3'��5' ��������: 5'��3' |
109 1 400 600 polA + + + |
120 1 ? 30 polB + + - |
175 ��10 10-20 30,000 polC + + - |
�� DNA�|�������[�[ II ���C���Ɋ֗^�i�Ȃ��Ă��זE�͎��ȂȂ��j�B
[DNA �|�������[�[I�̗��̍\��]DNA���Ɍ������Ă���l�q������Ɋ֗^���鉖��A�~�m�_�c���ԂŎ����B
 |
 |
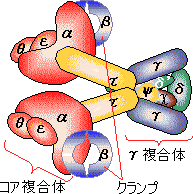
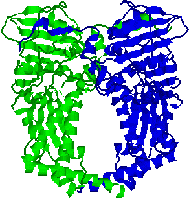
| [�咰��DNA�|�������[�[III�̍\��] | [b�T�u���j�b�g�̍\��] |
| aeq�F �R�A�����́i�G�}���S�j �@a�F DNA�|�������[�[ �@e�F 3'-5'�G�L�\�k�N���A�[�[ b�F �N�����v g�����́F �i�s�����i�i�N�����v�����j t�F �R�A�̓�ʑ̌`�����i |
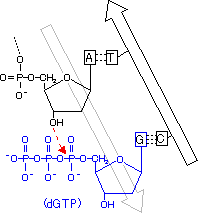
�@�咰��DNA�|�������[�[III��b�T�u���j�b�g��2�ʑ̂ŁC�h�[�i�c������Ă���12�{��a-�փ��b�N�X�łł���������35Å�̌���DNA��ʂ��ĕ�������DNA���O��Ȃ��悤�ɂ��邽�߁A�N�����v�ƌĂ��DNA�|�������[�[�̃R�A�y�f�̕������x��1000�{�ɍ��߂�B |
 �^�j�זE�̍y�f
�^�j�זE�̍y�f
| ���� | a | b | g | d | e |
| ���� ���q����(kD) �T�u���j�b�g ��������: 5'��3' ��������: 3'��5' ��������: 5'��3' �����ɕK�v�Ȃ��� RNA�v���C�}�[ DNA�v���C�}�[ �v���C�}�[�[�̌��� �A�t�B�W�R�������� �iDNA�����j�Q���j |
�j 120-220 4 + - - + + + + |
�j 30-50 1 + - - - + - - |
�~�g�R���h���A 150-300 2 + + - - + - - |
�j 140-160 2or3 + + - + + - + |
�j ? 1? + + - ? + - + |
[�q�gDNA�|�������[�[b�Ɛ�ڂ̂���DNA�̕�����]
�@�̂点���a-helix�C�̖���b-�\���ł��顒����ɏォ�猩���C��ڂ̂���DNA�̓�d�点�ʂ��Ă���B�|�������[�[b��DNA�������ݍ��ނ悤�Ɍ�������B
  
|
 DNA�̔��ۑ��I�����Ɋւ��鏔���
DNA�̔��ۑ��I�����Ɋւ��鏔���
 �����̕��@
�����̕��@(A) 2��������(q�^)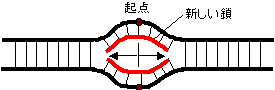 |
| �i�Q�{���̈�N�_���痼�����L���j |
(B) 1�������� |
| �i��N�_���������L���j |
| (C) ���[�����O�T�[�N���^ |
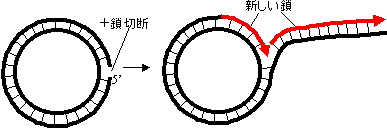 |
| [DNA�̔��ۑ��I����] �iA�j��ʂ̏ꍇ�A�iB�j�A�f�m�E�B���X�̏ꍇ�A�iC�j�t�@�[�WfX174�̏ꍇ |
 �����̊J�n�_�i�����N�_ replication origin�j
�����̊J�n�_�i�����N�_ replication origin�j| AATTTCGTCAAAAAATGCT�c�c�cATTTAAGTATTG�c�c�cTGAAAAGCAAGCA�c�c �c�cCTAAACATAAAATCT�c�c�c |
| [�y��̕����N�_(ARS)] A��T�ɕx�ށB�ԂŎ����z�K�{�ŁA�������͂��̍�p������B |
 DNA�̒����Z�����ǂ�����Ăق����̂��H
DNA�̒����Z�����ǂ�����Ăق����̂��H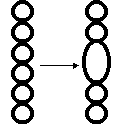
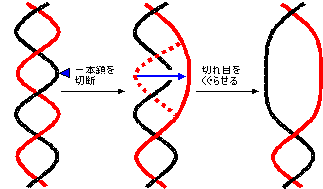
 |
| [�q�g�̃g�|�C�\�����[�[I/DNA������] |
 ���M���O(lagging)���͂ǂ̂悤�ɂ��č�������邩�H
���M���O(lagging)���͂ǂ̂悤�ɂ��č�������邩�H
 DNA�|�������[�[���Ԉ�����k�N���I�`�h��������������ǂ����邩�H
DNA�|�������[�[���Ԉ�����k�N���I�`�h��������������ǂ����邩�H
  
|
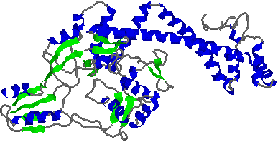  |
|
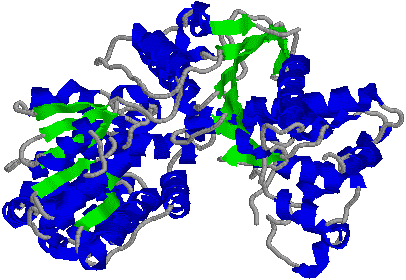
| [�W���C���[�XA�̍\��] | [�W���C���[�XB�̍\��] |
| �W���C���[�X�͒ʏ�A2B2�̎l�ʑ̂��`������B | |
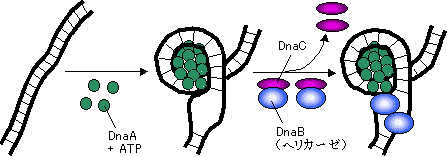 |
| [�咰�ۂ̕����N�_�ɂ����郉�Z���̊����߂�] |
 |
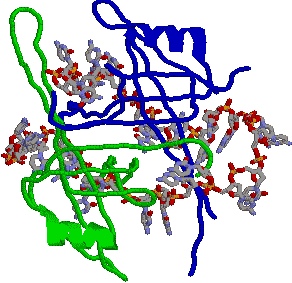
| [�w���J�[�[�̍\��] |
| ���q�̉����Ɏ߂ɐ[���a�����顂�����DNA���������� |
 |
| �܂�Ȃ������P�{��DNA�Ɍ�������SSB |
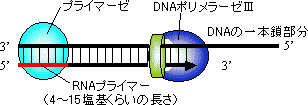 |
| [Leading���̍���] �ΐF�̓N�����v�ib�T�u���j�b�g�j |
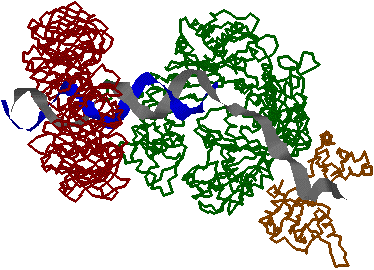 |
| [DNA�|�������[�[�V�̐G�}�@�\] |
| ���i�ԁj�̓N�����v�C�����i�Z�j�̓|�������[�[�̐G�}�T�u���j�b�g�C�E�i���j�͈�{��DNA�����^���p�N��(SSB)�ł���B�����ɂ͓�{���ƈ�{����DNA��������B |
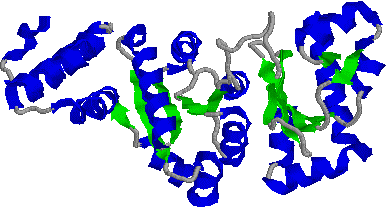 |
| [�咰�ۃv���C�}�[�[�̍\��] |
[lagging���̍���]
pol I 5'��3'���Ǹڱ��ފ�����RNA������ pol I 5'��3'����װ�ފ�����DNA�������� DNA���K�[�[ ��ڂ��Ȃ�
�咰�ۂɂ�����DNA�����̑S�̗̂���������B
  
|
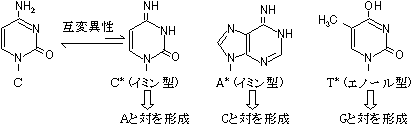
[DNA�|�������[�[�̍Z���@�\] C�̌ݕψِ���(C*)��A��F�� C�̌ݕψِ���(C*)�����܂���A�Ɖ�����`�����ADNA�|�������[�[�őg�ݍ��܂�� DNA�������ĊJ���� C*������C�ɖ߂�ƁA
C�EA�̃~�X�}�b�`��
������>DNA�|�������[�[������3'��5'�G�L�\�k�N���A�[�[�������~�X�}�b�`�k�N���I�`�h���o�� ������3'���[���������Ă��Ȃ��̂ŁA��������~����
  
|
 �@
�@
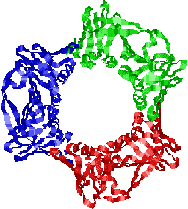 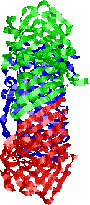 |
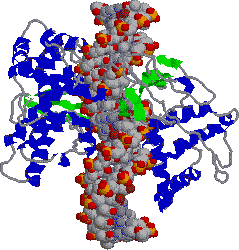
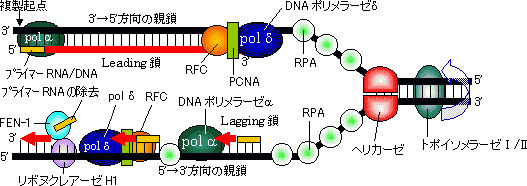
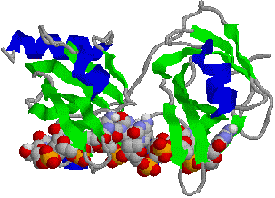
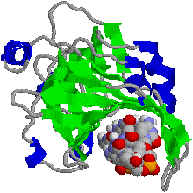
�@1���\�����قƂ�ǎ��Ă��Ȃ��ɂ��ւ�炸�C�q�g�̃N�����v�iPCNA�j���咰��DNA�|�������[�[III��b�T�u���j�b�g�Ƃ�������̍\�������Ă���B�����ɂ��������a-�փ��b�N�X�ň͂܂ꂽ�a35Å�̌�������CDNA�|�������[�[d��DNA�ɌŒ肷����������B�}�ŐF���������悤�ɁC���̃^���p�N���͂R�ʑ̂ō\������Ă��顃|�������[�[a�ƈ���āC�|�������[�[d������DNA�����ł���̂͂��̃^���p�N���̂������ł��� |
| [�q�gPCNA (proliferating cell nuclear antigen)]�エ��щ����猩���} | |
| �y�q�g�̍זE��DNA�z 23�A46�{�̐��F�́i2n�j DNA���̒�����99cm/haploid (n) 29�������/haploid S���̒�������10���� �����̑�������50�����/�b �i�咰�ہF500�����/�b�j DNA�̕����N�_����100����/���F�� |
 �@�q�g�̃Q�m��DNA�͒����i��̘g���Q�Ɓj�B
�@�q�g�̃Q�m��DNA�͒����i��̘g���Q�Ɓj�B
  
|
�@DNA�����j�Q�܂Ƃ��ẮADNA�|�������[�[�ڑj�Q������́A�����Ɋւ�鑼�̍y�f��j�Q������́ADNA��{���ɋ����������ĕ�����j�Q����Ȃǂ�����B�R���������B
�j�Q�� ��p �A���r�m�k�N���I�V�h
2',3'-�W�f�I�L�V�k�N���I�`�h
�@�O�����_(ddNTP)
�A�t�B�f�B�R�����זE����5'-�O�����_������ADNA�|�������[�[����
��j�Q�B
�咰��DNA�|�������[�[I��j�Q�i���I���@�ւ�
���p�j�B
dCTP��dTTP�Ɲh�R����DNA�|�������[�[a��j�Q�B2'-�f�I�L�V-2'-�A�W�h�V�`�W��
�@�O�����_
�m�{�r�I�V���A�i���W�L�V���_
�x���j�[���AHOE13548�v���C�}�[�[��j�Q�B
�咰�ۃg�|�C�\�����[�[II��j�Q�B
�g�|�C�\�����[�[I��j�Q�B�A�h���A�}�C�V���A�_�E�m�}�C�V��
�L���G�`�W�E��
�@DNA�̉���ΊԂɂ͂܂荞�ށi�C���^�[�J���[�V�����j�B
DNA��RNA������j�Q�B
�~�g�R���h���ADNA�|�������[�[g��j�Q
�i�C���^�[�J���[�V�����j�B
  |
�e�����A�̍\��
- �@������F�̖̂��[���e�����A(telomere)�Ƃ����B�e�����A�͌ŗL�̉���z���x���J��Ԃ����\�������Ă��顂��Ƃ��q�g�ł͂s�s�`�f�f�f�C�e�g���q���i�ł͂s�s�f�f�f�f��1000��ȏ�J��Ԃ��Ă��顂܂��C���̍��̖��[��12�`16����قǓˏo���Ă��顃e�����A�ɂ�TRF1��TRF2�Ƃ������C���L�̃^���p�N�������̂���������T-loop�\�����Ƃ�C�ˏo�������[�����艻���Ă���
J. D. Griffith et al., Cell (1999) 97, 503-514
�e�����A�͍זE�̎��������߂鎞�v
- �@�e�����A�����钷���܂ŒZ���Ȃ�ƁA�זE�͂���ȏ�זE���ł��Ȃ��Ȃ�B�e�����A�͍זE�̎��������߂鎞�v�̖�ڂ��ʂ����Ă���B�q�g�̎���������Ɗ֘A���Ă��邱�Ƃ���������Ă���B
�^�j�����͒�����DNA�������Ƃň�`�q�̑����g������e�Ղɂ��A��`�q�����ǂ���d�g�݂��B�����������ɁA����ꂽ������w���������ƂɂȂ�B�@����DNA�̕����̂��тɃe�����A�͒Z���Ȃ�i�e�����A����j
- �@leading�����DNA�|�������[�[�͍������I������Ɨ���Ă��܂����A���̎��Alagging���̕��̐��F�̖��[�̃e�����A�̈�̈ꕔ�͂܂������Ɏ肪���Ă��Ȃ��B�]���āA�����̂��т�lagging����5'�[�͒Z���Ȃ��Ă����B�܂��Aleading����5'�[��RNA�v���C�}�[����ŕ��������̂ŁA��������Z���Ȃ�B
[�^�j�זE�̒�����DNA�����ɂ�����e�����A���] �@�e�����A�̉���
- �@�Z���Ȃ��������̃e�����A�����̓e�������[�[�ʼn��������B�������A�q�g�̏ꍇ�A���̋@�\�͐��B�זE�₠���̊��זE�Ɍ��肳���B�̍זE�ɂ̓e�������[�[����������Ă��Ȃ����߁A�����̂��т�lagging���̒Z�k���N����B
[�e�������[�[�ɂ��e�����A�̉����@�\]
lagging���͉������ꂽ�e���𒒌^�Ƃ��Ă���ɐL�����

|