
長期的に見た場合、遺伝子の変化は環境に適応するために必要である(分子進化)。しかしながら、個体の生存といった短期的な時間内では、遺伝子の変化は個体の生存そのものを脅かす可能性(例えば発癌など)が考えられ、好ましいものではない。アミノ酸残基の変異が許容される程度はタンパク質によって大きく異なる。
| 各種タンパク質のアミノ酸配列の変化 |
| タンパク質 |
単位進化時間(百万年)* |
フィブリノペプチド
ヘモグロビン
シトクロム c
ヒストンH4 |
1.2
6.1
21
600 |
|
| * タンパク質中の100残基ごとに、1個の死に至らない変異が出現するのに要する平均時間。 |
フィブリノペプチドのように変異に要する時間の短いものもあれば、ヒストンH4(ヌクレオソームを構成するタンパク質)のようにほとんど変異を容認しないタンパク質もある。
従って、このようなタンパク質の遺伝子は正確な保全が要求される。これを支えるものとして、次のような機構がある。
 DNAの正確な複製
DNAの正確な複製
- Cの互変異性はC:G→C*:Aの変化をおこし、DNA複製のエラーを誘う。しかし、DNAポリメラーゼの校正機能により,複製のミスは108〜1010に1回程度に抑えられる。
 DNAの修復(DNA repair)
DNAの修復(DNA repair)
- DNAの鎖を修復・伸長するのは大腸菌ではDNAポリメラーゼ I であるが、真核細胞では多くのDNAポリメラーゼが関与する。
DNAは決して安定な化合物ではない。細胞内の反応性環境、毒物、紫外線や
放射線などによって、絶えず損傷を受けている。DNAの損傷は 細胞の老化や死、癌細胞の発生などを引き起こす原因となる。DNAの損傷には、次のような様々なタイプがある。
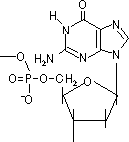
 脱プリン反応: H+によるアデニン、グアニンの切断
脱プリン反応: H+によるアデニン、グアニンの切断
 |
 |
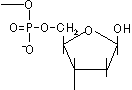 |
[自然に起きる脱プリン反応]
20,000ヶ所/ゲノム/日 |
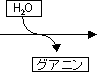
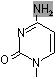
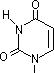
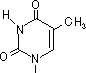
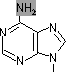
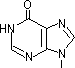
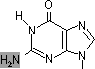
 脱アミノ反応: 水によるNH2→C=Oの変化
脱アミノ反応: 水によるNH2→C=Oの変化
 |
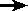
H2O
|
 |
|
 |
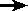
H2O
|
脱アミノしない |
| シトシン |
|
ウラシル |
チミン |
|
 |
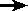
H2O
|
 |
 |
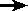
H2O
|
 |
| アデニン |
ヒポキサンチン |
グアニン |
|
キサンチン |
[自然に起きる脱アミノ反応]
200ヶ所/ゲノム/日 |
【DNAにウラシルを用いない理由】
シトシンから脱アミノ反応でウラシル(U)が生じる。UはAと対合するので,C→Uの変化は突然変異の原因となる。強い脱アミノ化剤である亜硝酸は突然変異を誘発する。 |
|
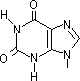
 チミン2量体(ダイマー)生成: 紫外線による損傷
チミン2量体(ダイマー)生成: 紫外線による損傷
 |
紫外線
 |
 |
光回復酵素
+
可視光 |
[紫外線によるチミンダイマー(シクロブタン環)
の生成と光回復酵素による修復] |
 アルキル化: アルキル化剤→プリン塩基のN,O原子をアルキル化。
アルキル化: アルキル化剤→プリン塩基のN,O原子をアルキル化。
- S-アデノシルメチオニン→グアニンのN7をメチル化。
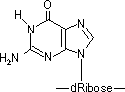 |
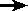 |
 |
 |
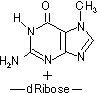 |
[自然に起きるグアニンのアルキル化]
230,000ヶ所/ゲノム/日 |
 活性酸素による酸化: ・OHラジカル→グアニンのC8-HをC-OHに変化
活性酸素による酸化: ・OHラジカル→グアニンのC8-HをC-OHに変化
- [170ヶ所/ゲノム/日]
真核細胞のDNAポリメラーゼa,d,e,gは核やミトコンドリアのDNA複製に関与するが,a,g以外は修復も行う。
DNA修復においてはDNAポリメラーゼb以外に,多くのDNAポリメラーゼが関わっていることが最近,明らかにされた。もっぱら修復に関わる酵素はいずれもDNA鎖伸長能
(processivity) が低く,また,忠実度 (fidelity) の低いものが多い。ただし,DNAポリメラーゼhはチミン2量体を正確に修復する。
DNA修復に関与するヒトのDNAポリメラーゼ
| DNAポリメラーゼ |
遺伝子 |
機能 |
pol b
pol g
pol d
pol e
pol z
pol h
pol q
pol i
pol k
pol l
pol m
deoxycytidyl
transferase |
POLB
POLG, POLG2
POLD1, POLD2
POLE, POLE2
POLZ
POLH
POLQ
POLI
POLK
POLL
POLM
REV1
|
塩基除去修復
ミトコンドリアDNA複製と修復
DNA複製とヌクレオチド除去修復
DNA複製とヌクレオチド除去修復
損傷乗り越え修復,組換え修復
紫外線損傷乗り越え修復
DNA修復?
損傷乗り越え修復?
損傷乗り越え修復?
減数分裂での修復?
体細胞突然変異?
損傷乗り越え修復 |
 除去修復
除去修復
 ヌクレオチド除去修復(nucleotide excision repair, NER)
ヌクレオチド除去修復(nucleotide excision repair, NER)
- 1. 損傷部位は通常,真核細胞ではXPC-HR23B複合体が認識するが,転写中はRNAポリメラーゼ
II が認識する(下記の転写共役DNA修復を見よ)。次いで,TFIIH(ヘリカーゼ), XPG, XPA, RPAタンパク質群が損傷部位の2本鎖DNAのらせんをほどく。
2. XPE-ERCC1複合体とXPGが損傷を含むオリゴヌクレオチド(約25-30塩基)をDNAから切り取る。
3. 生じた1本鎖の隙間を原核細胞ではDNAポリメラーゼ Iが,真核細胞ではPCNA,RF-Cと共同して,DNAポリメラーゼdとeが埋める。チミン2量体の修復も可能。
4. リガーゼがDNA鎖をつなぐ。

1.認識
|
|

2.切れ目を
挿入 |
|

3.DNA切り
取りと修復
合成 |
|

4.リガーゼで
連結 |
 塩基除去修復(base excision repair, BER)
塩基除去修復(base excision repair, BER)
- 変異塩基をDNAグシコシラーゼが切り取る。生じた脱プリン/ピリミジン部位(AP部位という)のDNA1本鎖をAPエンドヌクレアーゼが切る。以後、DNAポリメラーゼbが1塩基ギャップを生じて修復される経路か,DNAポリメラーゼdとeが比較的長い修復を行った後,FEN 1がはねだされた損傷を含むオリゴヌクレオチドを切り取って修復を行う経路に分かれる。

変異塩基
|
グリコシラーゼ
で異常塩基を
切り取る |

AP部位 |
|
AP部位を
APエンド
ヌクレアーゼ
で切る |
 |
1.
1塩基ギャップ

|
1塩基ギャップを生じて修復
2. ヌクレオチド除去修復と似た
経路のどちらかで修復合成し,
リガーゼがDNA鎖をつなぐ。 |
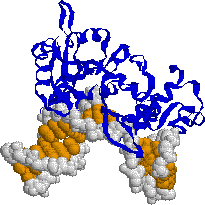 |
| ニック(切れ目)の入ったDNAを修復中のDNAポリメラーゼb |
 複製後修復(損傷を抱えたDNAが複製に入った場合に適用される)
複製後修復(損傷を抱えたDNAが複製に入った場合に適用される)
 相同DNA組み換え修復
相同DNA組み換え修復
- 損傷のない他方の鎖の組換えで損傷部分を補充した後,損傷のないDNAに生じた隙間を埋めて閉じる。
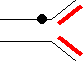
1.損傷と複製
|
|
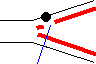
2.娘鎖に隙間を
残したまま複製
|
|
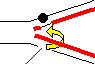
3.娘鎖にある同じ
配列を借用
(組換え) |
|

4.親鎖の隙間を修復
(修復合成) |
 損傷乗り越え修復
損傷乗り越え修復
- 紫外線によりチミン2量体が生じた場合,上記のヌクレオチド除去修復で修復されるが,除去されない場合はDNA複製が停止する。そこで,真核細胞の場合DNAポリメラーゼh が正しい塩基(アデニン)を挿入する。DNAポリメラーゼz もチミン2量体を修復するが,しばしば誤った塩基を挿入する(誤りがちな乗り越え修復)。
大腸菌ではDNAポリメラーゼV (以前はUmuD'2と呼ばれていたが,最近,DNAポリメラーゼ活性をもつことが分かりpol Vと命名された)が行う。[なお,大腸菌には忠実度の低いDNAポリメラーゼIVも知られていて,適応変異という別の修復に関与する]。
 ミスマッチ修復(mismatch repair, MMR)
ミスマッチ修復(mismatch repair, MMR)
- 1. DNA複製におけるエラーでミスマッチが起き,DNAポリメラーゼによる校正がなされなかった場合,新生の娘鎖のミスマッチを大腸菌ではMutS-MutL複合体,ヒトではhMSH2-hMSH6複合体かhMLH1-hPMS2複合体が認識する。
2. 大腸菌では非メチル化鎖を新生鎖と見なしてMutHタンパク質が損傷部位に切れ目を入れた後,エクソヌクレアーゼでミスマッチ塩基を取り除く。
3. 真核細胞ではミスマッチ塩基を含むオリゴヌクレオチドがエンドヌクレアーゼで除去され,PCNA,RF-Cと共同して,DNAポリメラーゼdとeが隙間を埋める。
 誘導SOS修復(大腸菌)
誘導SOS修復(大腸菌)
- 大腸菌が変異原に曝された時に起きる防御機構である。DNAに大きな損傷が起きると、生じた1本鎖DNAにRecAタンパクが結合し,SOSボックス(オペレーター部位)に結合しているLexAリプレッサータンパクを不活性化する。その結果、SOS遺伝子の転写が開始されてDNAポリメラーゼVとRecAタンパク質を生成する。これらが傷を修復する。SOS修復系は元の塩基がわからなくても適当な塩基を入れるので変異原性(誤りがちな修復,error-prone repair)である。

1.近接した複数
の損傷 |
|

2.損傷部位全域
の切り取り |
SOS
誘導
|

3.SOS修復系で
修復合成。
この時,相手の
鎖の損傷部に
は適当な塩基
が入れられる。 |
 光回復
光回復
- 光回復酵素(フォトリアーゼ), FADH2, プテリンと300-500nmの光によりシクロブタン環を開裂。
 適応修復(adaptive repair)
適応修復(adaptive repair)
- グアニンのメチル化で生じるO6-メチルグアニンは、O6-メチルグアニンDNAアルキルトランスフェラーゼで脱メチル化される。このSH酵素はメチル化されて失活する(自殺酵素)。
 転写共役修復(transcription-coupled repair, TCR)
転写共役修復(transcription-coupled repair, TCR)
- DNAが転写されている時に損傷部位に遭遇した場合,転写共役DNA修復が発動される。真核生物では,RNAポリメラーゼ
II が損傷を認識し,XPAタンパク質をはじめとして多くのタンパク質が関与するが,詳細は不明である。修復はDNAポリメラーゼdとeが行う。この修復系は他の修復より格段に高速である。







 DNAの正確な複製
DNAの正確な複製
 DNAの修復(DNA repair)
DNAの修復(DNA repair)


 脱プリン反応: H+によるアデニン、グアニンの切断
脱プリン反応: H+によるアデニン、グアニンの切断 脱アミノ反応: 水によるNH2→C=Oの変化
脱アミノ反応: 水によるNH2→C=Oの変化 チミン2量体(ダイマー)生成: 紫外線による損傷
チミン2量体(ダイマー)生成: 紫外線による損傷 アルキル化: アルキル化剤→プリン塩基のN,O原子をアルキル化。
アルキル化: アルキル化剤→プリン塩基のN,O原子をアルキル化。
 活性酸素による酸化: ・OHラジカル→グアニンのC8-HをC-OHに変化
活性酸素による酸化: ・OHラジカル→グアニンのC8-HをC-OHに変化





 除去修復
除去修復
ヌクレオチド除去修復(nucleotide excision repair, NER)
塩基除去修復(base excision repair, BER)
 複製後修復(損傷を抱えたDNAが複製に入った場合に適用される)
複製後修復(損傷を抱えたDNAが複製に入った場合に適用される)
相同DNA組み換え修復
損傷乗り越え修復
polz
ミスマッチ修復(mismatch repair, MMR)
誘導SOS修復(大腸菌)
 光回復
光回復
 適応修復(adaptive repair)
適応修復(adaptive repair)
 転写共役修復(transcription-coupled repair, TCR)
転写共役修復(transcription-coupled repair, TCR)
